A estas alturas de la pandemia todos hemos oído hablar de la técnica de la PCR. ¿Sabemos qué es una PCR y cuándo se utiliza? ¿Tenemos claro cuáles son los errores más comunes que pueden surgir?
A estas alturas de la pandemia todos hemos oído hablar de la técnica de la PCR. A muchos de nosotros nos han realizado la prueba o, al menos, todos tenemos algún familiar o amigo que se ha sometido al test. Pero, ¿sabemos qué es una PCR y cuándo se utiliza? ¿Tenemos claro cuáles son los errores más comunes que pueden surgir?
Empecemos por aclarar que la PCR es una técnica de biología molecular que se lleva aplicando en el campo de la investigación desde hace más de tres décadas. Desarrollada en 1986 por el bioquímico estadounidense Kary Mullis, se basa en una reacción enzimática mediada por la polimerasa. De ahí su nombre, derivado de las siglas en inglés de Polymerase Chain Reaction (Reacción en Cadena de la Polimerasa, PCR).
La polimerasa es una proteína que está presente en todas las células de cualquier ser vivo y se encarga de duplicar el material genético –el ADN– en los momentos previos a la división celular. Con la PCR aprovechamos esta habilidad de la enzima para realizar muchas copias de un fragmento concreto de ADN.
¿Cuándo se utiliza la PCR?
La PCR es útil cuando somos capaces de indicar a la enzima polimerasa qué fragmento exacto de ADN es el que queremos que copie. Eso significa que para poder recurrir a ella debemos conocer previamente la estructura del material genético y tener claro el fragmento que nos interesa.
Entre otras cosas, sirve para:
detectar infecciones producidas por microorganismos patógenos, como el coronavirus Sars-CoV-2 o la gripe en humanos, y también Leishmania en perros;
saber cuánta cantidad de un microorganismo (bacteria, virus, hongo…) hay en un determinado alimento;
detectar si una bacteria posee genes específicos de resistencia a un antibiótico concreto;
medir los niveles de expresión de un gen y comprender por qué bacterias con el mismo código genético responden de manera diferente en función de las condiciones a las que son sometidas.
También se recurre a ella en medicina forense, para la identificación de víctimas o agresores. O en paleontología para identificar restos biológicos que no se pueden catalogar a simple vista.
¿Cuáles son los errores más comunes que pueden surgir?
Aunque valiosa, la PCR no es infalible. Para entender cómo de fiable resulta conviene tener claros los conceptos de sensibilidad (probabilidad de que una muestra positiva genere un resultado positivo) y especificidad (probabilidad de que una muestra negativa origine un resultado negativo).
En el caso de la PCR del coronavirus se estima que su sensibilidad oscila entre el 70% y el 90% y su especificidad se sitúa por encima del 99,5%. Esto implica que prácticamente no se producen falsos positivos (las muestras negativas apenas devuelven resultados positivos). Pero sí puede haber algún falso negativo (muestra positiva que da lugar a un resultado negativo).
En ambos casos, el error suele derivar de un fallo bien durante el propio procesado de la muestra o bien durante la realización de la prueba.
Para poder comprender por qué se producen este tipo de errores es esencial conocer cómo se ejecuta la propia técnica de la PCR y cuáles son los puntos críticos de todo el proceso, ya que un fallo en alguno de ellos podría derivar en un resultado erróneo.
Los principales puntos críticos del proceso son los siguientes:
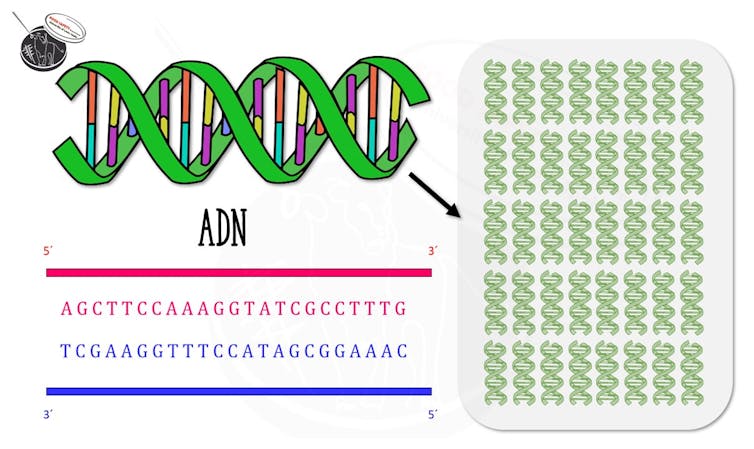
1. Elección del fragmento de ADN que queremos copiar
Es esencial definir qué fragmento exacto se va a copiar, de manera que si lo que queremos es detectar un microorganismo determinado en una muestra (como por ejemplo el coronavirus SARS-CoV-2 en una persona, o la bacteria Listeria monocytogenes en un alimento) lo que deberemos hacer es «decirle» a la polimerasa qué es lo que estamos buscando y cómo encontrarlo.
Para ello, hay que diseñar pequeños fragmentos de ADN –llamados cebadores o primers– que delimitan nuestro fragmento e indican a la polimerasa dónde empezar y terminar de copiar. Si queremos detectar el coronavirus deberemos copiar un fragmento totalmente diferente al que copiaríamos si quisiéramos detectar L. monocytogenes. Por lo tanto, habrá que diseñar cebadores específicos en cada caso.
2. Toma de la muestra
Siguiendo con el ejemplo de detección del coronavirus, resulta crucial recoger la muestra en la ubicación y momento adecuados. Se toman muestras nasofaríngeas porque es mucho más probable encontrar el virus en estas zonas que directamente en la cavidad bucal.
Pero también influye el momento en el que se recoge la muestra. Si la infección es demasiado reciente se puede obtener un resultado negativo, dada la baja carga viral en ese momento, aunque la persona acabe siendo positiva días después.
3. Obtención del ADN
Este paso consiste en llevar a cabo la extracción del material genético, es decir, retirar de la muestra todo aquello que no nos interesa. Por ejemplo los restos del hisopo con el que se ha tomado la muestra nasofaríngea, en el caso del coronavirus, o los restos del alimento que se está procesando, en el caso de L. monocytogenes.
Para ello, se deposita la muestra en algún diluyente líquido que facilite todo el proceso y se trata con calor para romper las células. Después se llevan a cabo varios ciclos de centrifugación que permiten separar la muestra en distintas fases, quedando el material genético suspendido en el diluyente.
Además, puede que el material genético de partida sea ARN. En esos casos habría que realizar un paso previo a la amplificación que consiste en la conversión de dicho ARN en ADN.
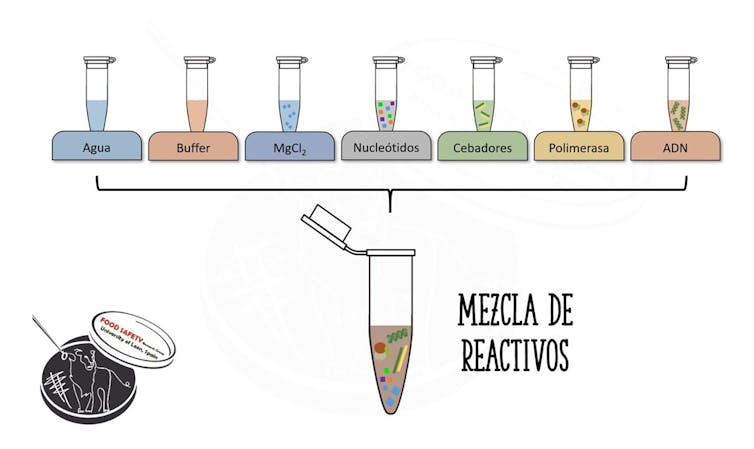
4. Adición de reactivos
Llegados a este punto es esencial elegir correctamente la cantidad de cada uno de los reactivos que se añade, pudiendo variar en función del fragmento que se quiera copiar. Los componentes principales de la reacción son los siguientes:
Agua: para tener un volumen final suficiente donde se desarrolle la reacción.
Buffer de reacción: para mantener unas condiciones de pH determinadas y continuas durante todo el proceso.
Iones de magnesio: para estabilizar la acción de la polimerasa y permitir que pueda hacer «su trabajo» sin problemas.
Nucleótidos (dNTPs): piezas que la polimerasa irá añadiendo al formar cada nueva copia de ADN.
Cebadores: pequeños fragmentos de ADN que se unen a los extremos de lo que queremos copiar y que sirven de molde para indicar a la polimerasa lo que reproducir.
Polimerasa: enzima que realiza la reacción de amplificación.
Muestra: el ADN que hemos extraído previamente.
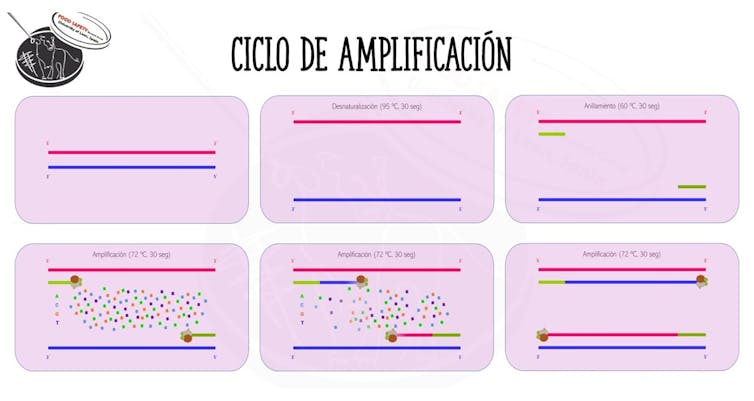
5. Reacción de amplificación
El siguiente momento crítico se produce durante la propia reacción de amplificación del fragmento. Se trata de la repetición sucesiva de 3 simples pasos, que conforman un ciclo de amplificación:
Desnaturalización. Separación de las 2 hebras que conforman el ADN mediante la aplicación de calor (95 ºC).
Anillamiento. Colocación de los cebadores en la posición adecuada para marcar el fragmento a copiar. La temperatura debe descender aproximadamente a 60 ºC.
Elongación: Adición de los nucleótidos, uno a uno, por parte de la polimerasa, desde la posición en la que se colocan los cebadores. Aquí la temperatura debe rondar los 72 ºC.
Cada vez que se termina un ciclo de amplificación, se duplica la cantidad de fragmento de ADN que teníamos en un principio. Si partimos de una única copia del fragmento y hacemos 20 ciclos de amplificación, al final de la reacción tendremos aproximadamente 1 millón de copias del fragmento inicial.
6. Revelado de los resultados
Finalmente, para saber si se han realizado correctamente las copias de nuestro fragmento, habría que revelar los resultados. Para ello, se pueden distinguir dos tipos básicos de PCR:
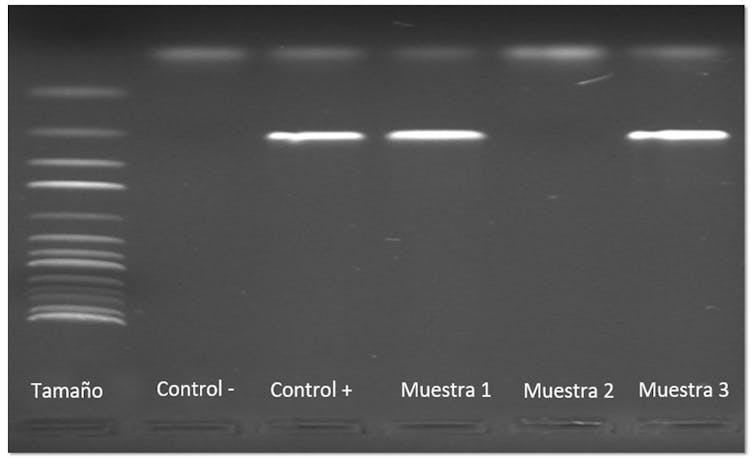
- PCR convencional: habría que revelar cada muestra analizada utilizando una técnica conocida como electroforesis en gel de agarosa. Esta herramienta nos permite separar los fragmentos copiados en función de su tamaño. Si la muestra es positiva a un microorganismo o tiene un gen que estamos buscando (por ejemplo, de resistencia a un determinado antibiótico), aparecerá una línea en el gel correspondiente a nuestro fragmento de interés. Si, por el contrario, la muestra es negativa, no se observará ninguna línea.
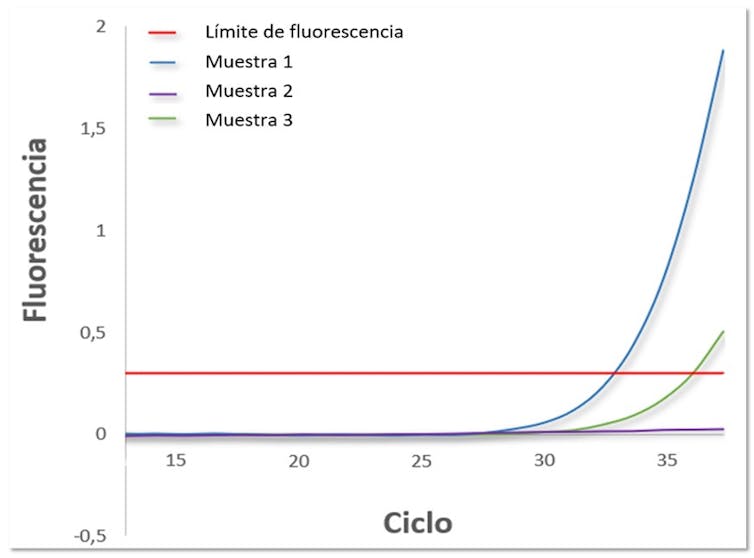
PCR en tiempo real: en este caso el proceso es ligeramente diferente, ya que habría que añadir un componente en el inicio de la reacción. Se trata de un compuesto, llamado fluorocromo, capaz de emitir fluorescencia cada vez que se hace una copia nueva de nuestro fragmento. Se necesita una máquina capaz de medir estos niveles de fluorescencia durante todo el proceso de amplificación. Las muestras serán positivas si superan un límite de fluorescencia preestablecido.
Esta técnica también permite determinar la concentración que hay, en cada muestra, de un determinado microorganismo, en función de cuántos ciclos de amplificación tarde en superar dicho límite marcado. Cuanto mayor sea la concentración del microorganismo en la muestra, menor será el número de ciclos necesarios para la detección de la fluorescencia.
Parece que la pregunta que daba pie a este artículo, ¿cuándo es útil y cuándo no la prueba de la PCR?, no tiene una única respuesta válida. Se utilizará siempre y cuando se quiera copiar un determinado fragmento de ADN. Y, como hemos visto, esto puede aplicarse a multitud de situaciones diferentes (y no solo para diagnosticar infecciones por coronavirus).
Además, si queremos que su uso sea el correcto, habrá que tener en cuenta una serie de puntos críticos cuya alteración podría derivar en un resultado erróneo. Aun así, se trata de una prueba tan sensible y específica que estos errores son muy poco frecuentes.![]()
Sarah Panera Martínez, Contratada predoctoral - Seguridad Alimentaria y Microbiología de los Alimentos, Universidad de León; Carlos Alonso Calleja, Catedrático de Universidad, Área de Conocimiento de Nutrición y Bromatología, Facultad de Veterinaria, Universidad de León; Cristina Rodríguez Melcón, Contratada predoctoral - Seguridad Alimentaria y Microbiología de los Alimentos, Universidad de León; Rosa Capita González, Catedrática de Universidad, Área de Conocimiento de Nutrición y Bromatología, Facultad de Veterinaria, Universidad de León y Víctor Serrano Galán, Técnico de Apoyo a la Investigación, Universidad de León
Este artículo fue publicado originalmente en The Conversation. Lea el original.








![¡En plena audiencia! Acusado atacó a jueza que le negó la libertad condicional [VIDEO]](https://e.rpp-noticias.io/small/2024/01/04/mmd-vd-20240104-clj-usa-justiciacriminalidad-349g2vy-es-1520871mp4_1520872.webp)








